聚类热图的层级关系是固定的,但分支的左右镜像是可变的。如何让聚类结果更好的呈现我们想要的顺序呢?看下面的操作。
数据示例
exprTable <- read.table("exprTable.txt", sep="\t", row.names=1, header=T, check.names = F)
exprTable测试时直接拷贝这个数据即可
## Zygote 2_cell 4_cell 8_cell Morula ICM
## Pou5f1 1.0 2.0 4.0 8.0 16.0 32.0
## Sox2 0.5 1.0 2.0 4.0 8.0 16.0
## Gata2 0.3 0.6 1.3 2.6 5.2 10.4
## cMyc 10.4 5.2 2.6 1.3 0.6 0.3
## Tet1 16.0 8.0 4.0 2.0 1.0 0.5
## Tet3 32.0 16.0 8.0 4.0 2.0 1.0绘制一个聚类热图很简单
library(pheatmap)
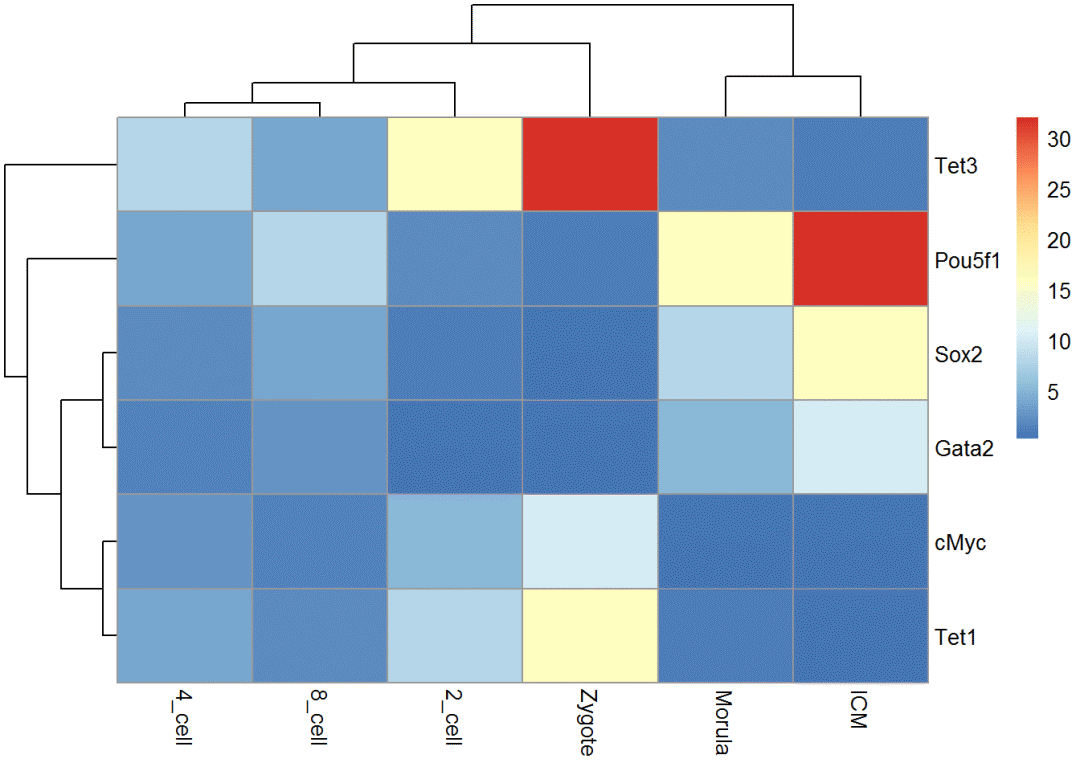
pheatmap(exprTable)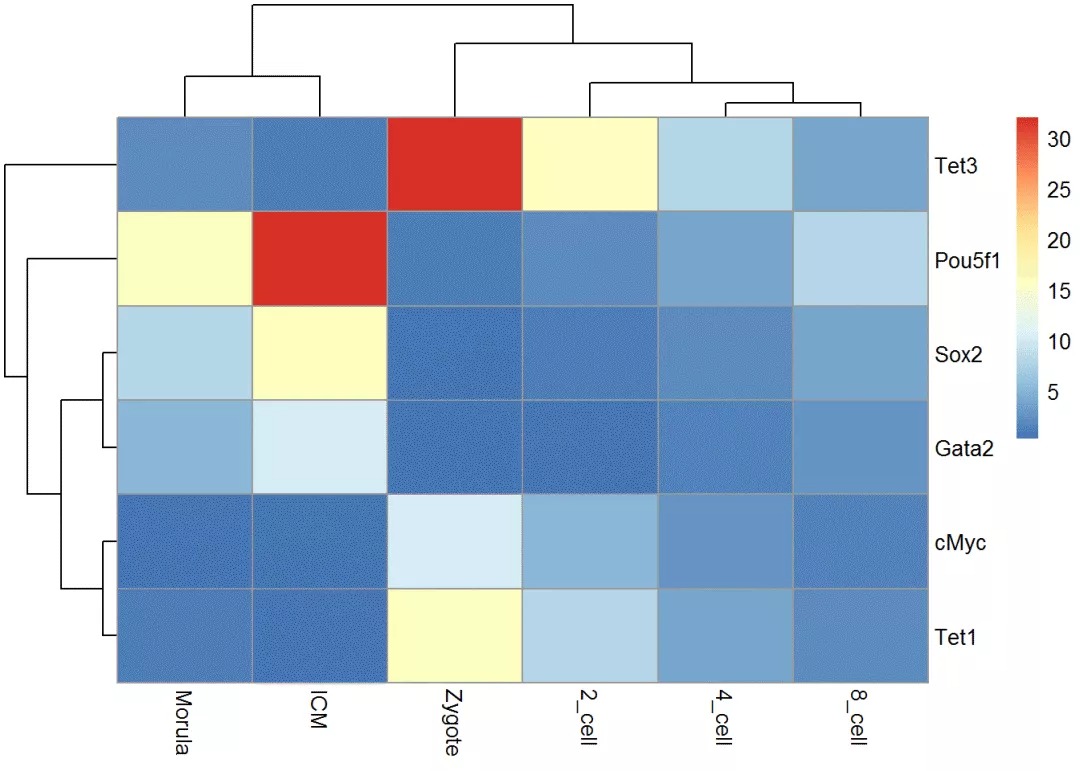
如何自定义分支顺序呢
自己做个hclust传进去,顺序跟pheatmap默认是一样的
exprTable_t <- as.data.frame(t(exprTable))
col_dist = dist(exprTable_t)
hclust_1 <- hclust(col_dist)
pheatmap(exprTable, cluster_cols = hclust_1)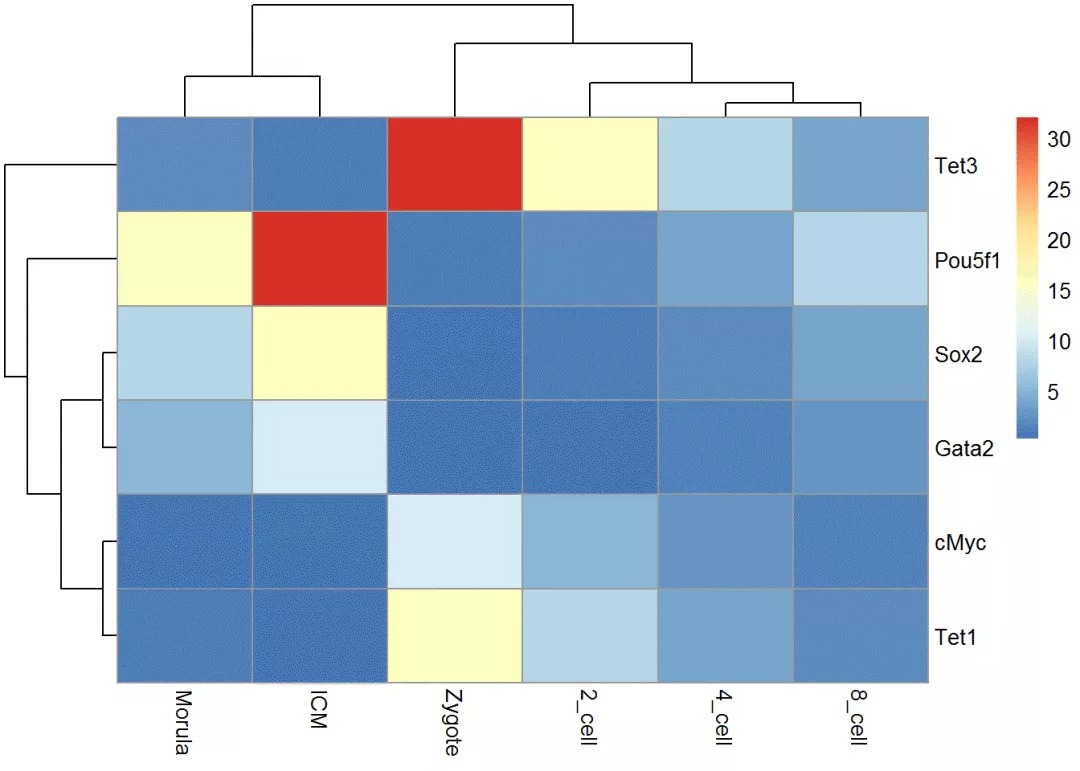
人为指定顺序排序样品
按发育时间排序样品
manual_order = c("Zygote", "2_cell", "4_cell", "8_cell", "Morula", "ICM")
dend = reorder(as.dendrogram(hclust_1), wts=order(match(manual_order, rownames(exprTable_t))))
# 默认为mean,无效时使用其他函数尝试
# dend = reorder(as.dendrogram(hclust_1), wts=order(match(manual_order, rownames(exprTable_t))), agglo.FUN = max)
col_cluster <- as.hclust(dend)
pheatmap(exprTable, cluster_cols = col_cluster)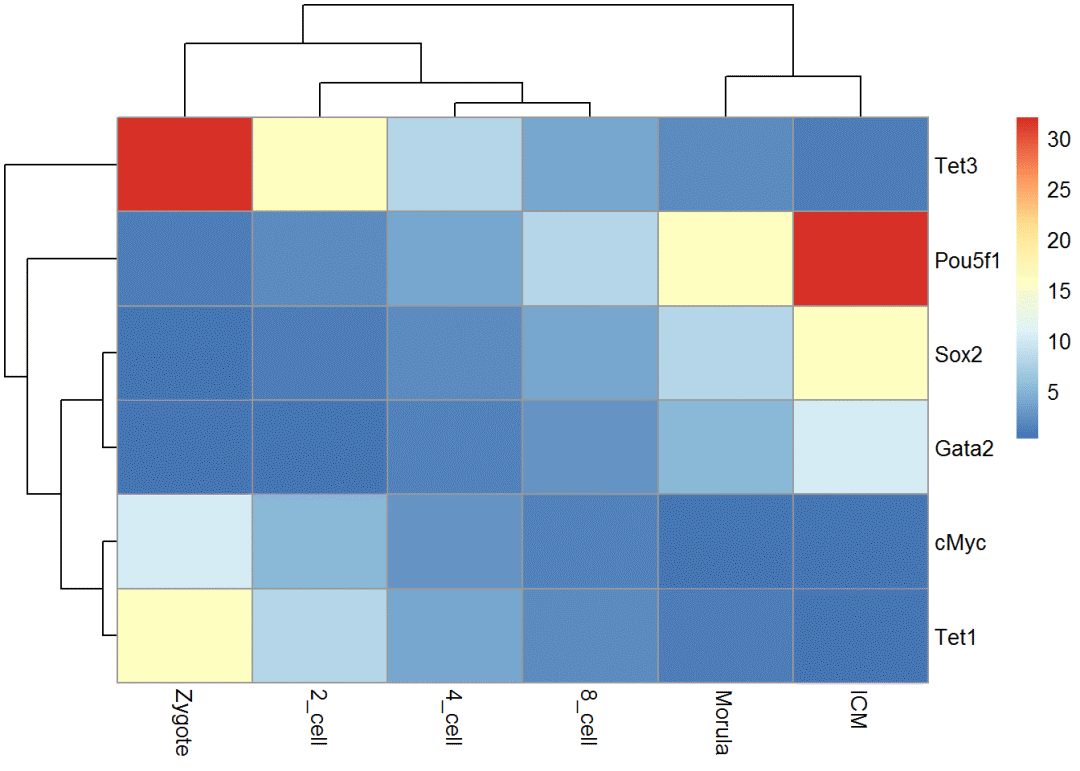
按某个基因的表达由小到大排序
可以按任意指标排序,基因表达是一个例子。
dend = reorder(as.dendrogram(hclust_1), wts=exprTable_t$Tet3)
col_cluster <- as.hclust(dend)
pheatmap(exprTable, cluster_cols = col_cluster)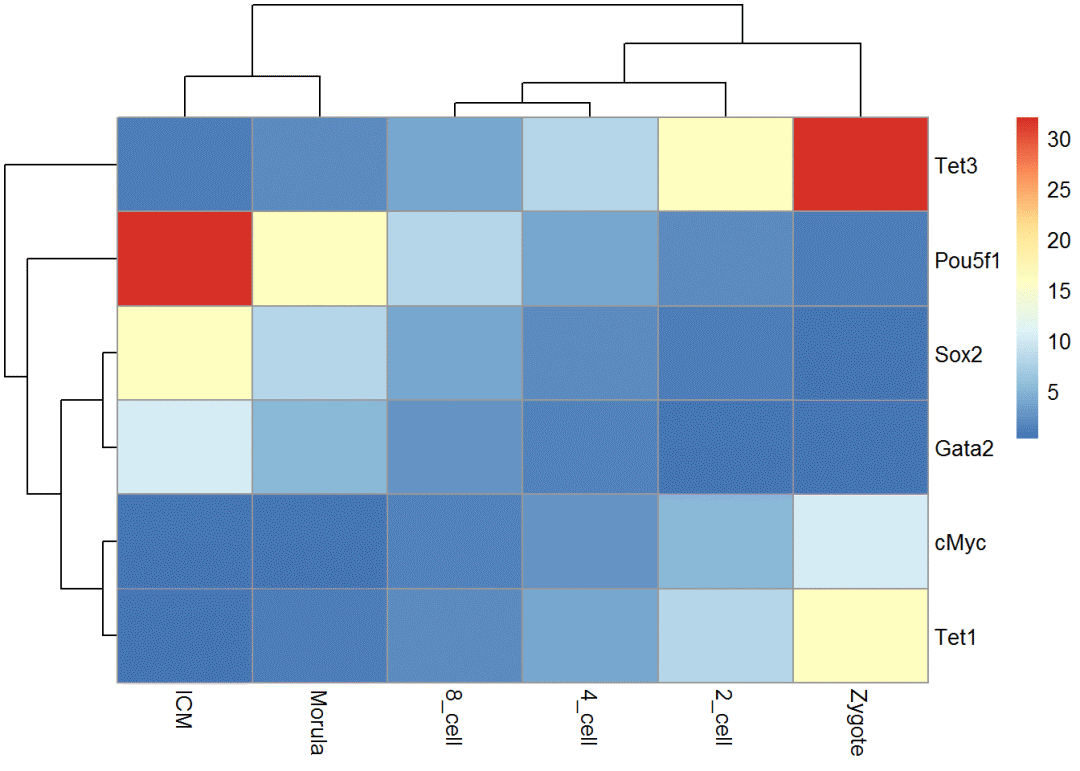
按某个基因的表达由大到小排序
dend = reorder(as.dendrogram(hclust_1), wts=exprTable_t$Tet3*(-1))
col_cluster <- as.hclust(dend)
pheatmap(exprTable, cluster_cols = col_cluster)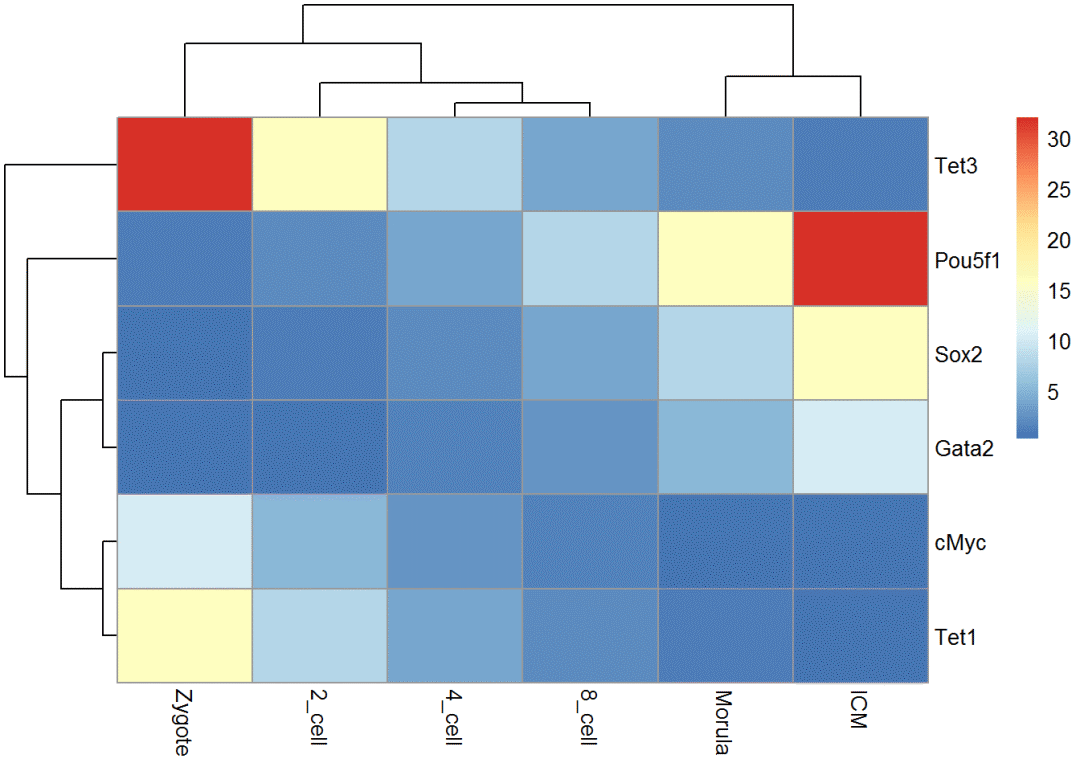
按分支名字(样品名字)的字母顺序排序
library(dendextend)
col_cluster <- hclust_1 %>% as.dendrogram %>% sort %>% as.hclust
pheatmap(exprTable, cluster_cols = col_cluster)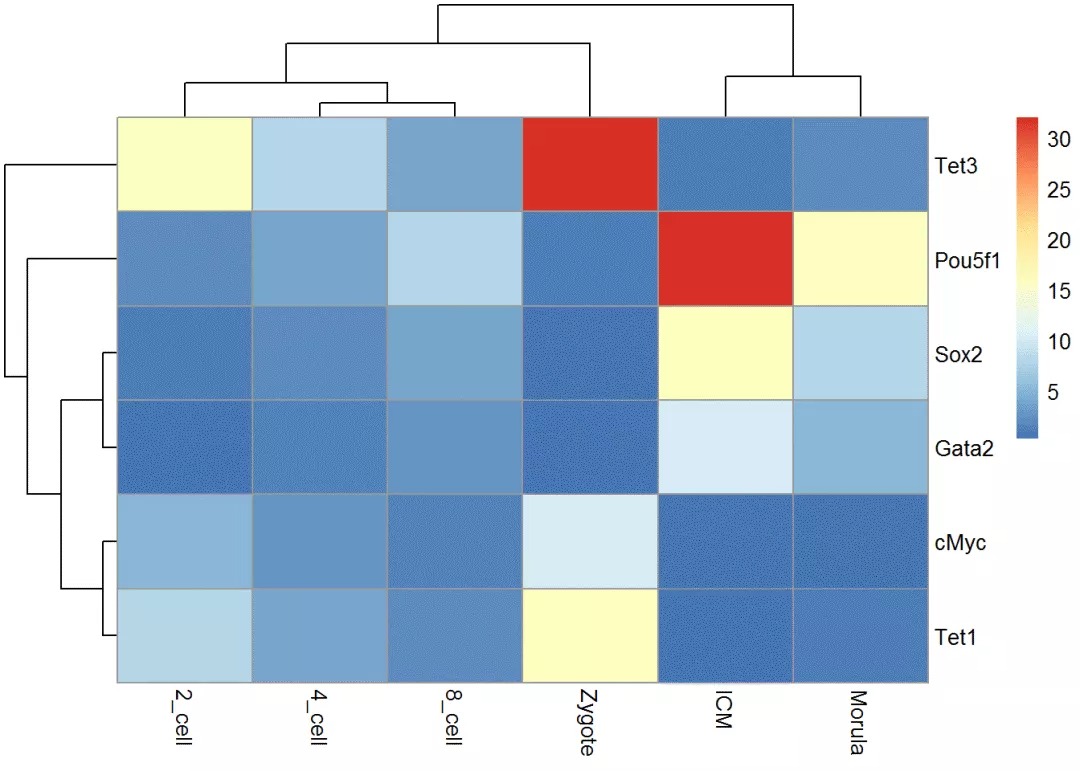
梯子形排序:最小的分支在右侧
col_cluster <- hclust_1 %>% as.dendrogram %>% ladderize(TRUE) %>% as.hclust
pheatmap(exprTable, cluster_cols = col_cluster)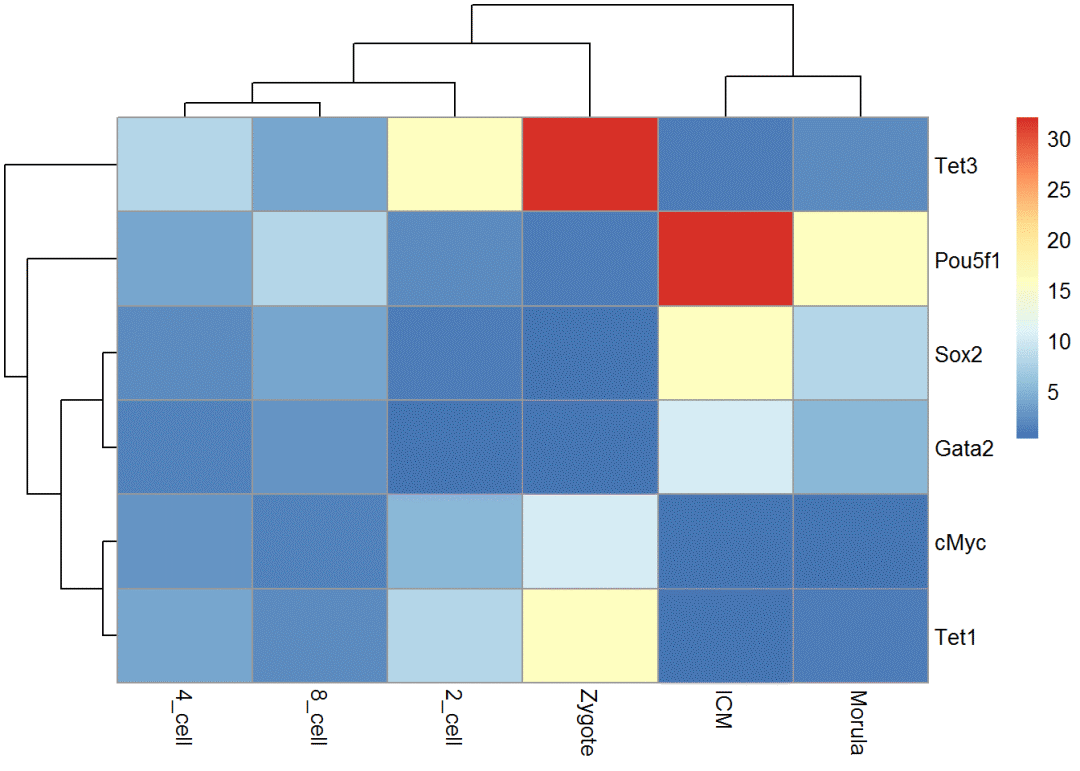
梯子形排序:最小的分支在左侧
col_cluster <- hclust_1 %>% as.dendrogram %>% ladderize(FALSE) %>% as.hclust
pheatmap(exprTable, cluster_cols = col_cluster)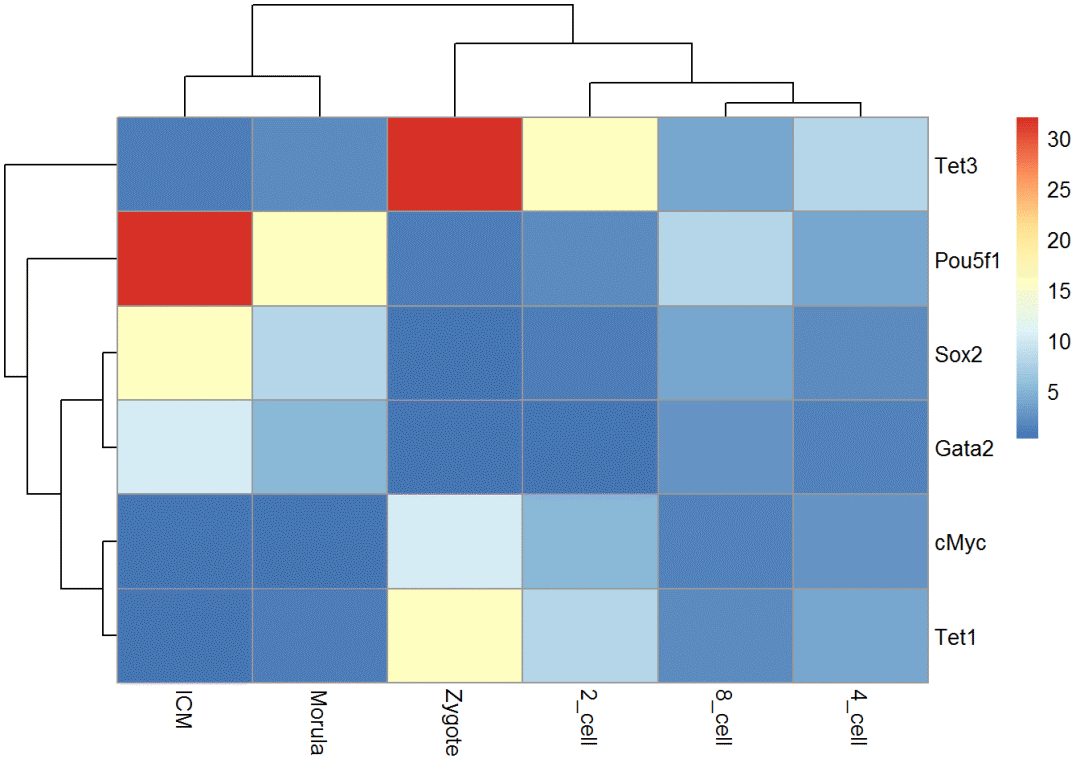
按特征值排序
样本量多时的自动较忧排序
sv = svd(exprTable)$v[,1]
dend = reorder(as.dendrogram(hclust_1), wts=sv)
col_cluster <- as.hclust(dend)
pheatmap(exprTable, cluster_cols = col_cluster)